Importando dados do UCINET
Em meu último post, trabalhamos a importação de dados a partir do software Pajek, ferramenta gratuita e disponível para usuários do sistema Windows. Para importamos dados do UCINET, outro software bastante comum para ARS, podemos gerar um arquivo .txt com o menu Data - Export - Raw Data tomando o cuidado de deixar as opções Field width = VARIABLE e a opção Separate numbers with = others (;) para estabelecer ponto-e-vírgula como separador. No R, a rotina consiste de ler os dados do arquivo .txt, transformá-los em um objeto de classe matrix e separar as redes (temos que lidar com cada uma em separado diferente do UCINET) para começar as análises. Podemos importar os dados com os comandos
rede <- read.csv2("C:/Users/.../Krack-High-Tec.txt", header = T, stringsAsFactors = F)
rede <- as.matrix(rede)
krack_advice <- rede[1:21,]
krack_friendship <- rede[22:42,]
krack_reports_to <- rede[43:63,]
Agora, transformamos as matrizes em objetos igraph.
library(igraph)
krack_advice <- graph_from_adjacency_matrix(krack_advice)
krack_friendship <- graph_from_adjacency_matrix(krack_friendship)
krack_reports_to <- graph_from_adjacency_matrix(krack_reports_to)
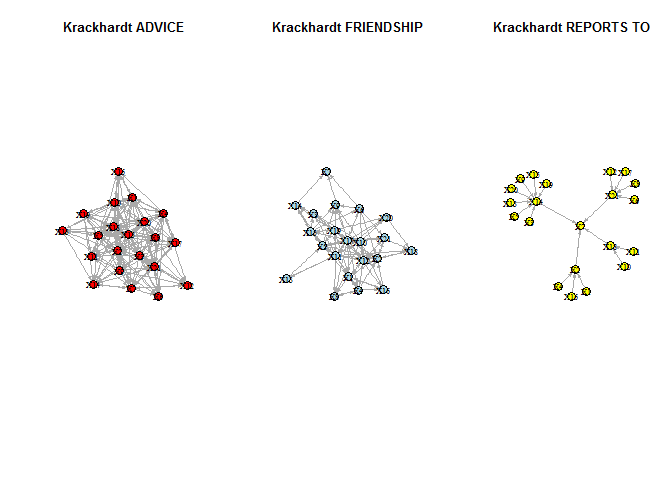
Para plotar as três ao mesmo tempo:
par(mfrow=c(1,3))
plot(krack_advice, vertex.label.cex=.8, vertex.label.color="black", vertex.color="red", edge.arrow.size=0.2, main="Krackhardt ADVICE")
plot(krack_friendship, vertex.label.cex=.8, vertex.label.color="black", vertex.color="lightblue", edge.arrow.size=0.2, main="Krackhardt FRIENDSHIP")
plot(krack_reports_to, vertex.label.cex=.8, vertex.label.color="black", vertex.color="yellow", edge.arrow.size=0.2, main="Krackhardt REPORTS TO")
Para importar os atributos presentes no arquivo High-Tec-Attributes, faça o mesmo processo para exportar os dados para uma Raw Matrix no UCINET e no R faça
atributos <- read.csv2("C:/Users/.../High-Tec_Attributes.txt", header = T, stringsAsFactors = F)
Para colocamos os vetores de atributos junto aos objetos igraph fazemos
V(krack_advice)$age <- atributos$AGE
V(krack_advice)$tenure <- atributos$TENURE
V(krack_advice)$level <- atributos$LEVEL
V(krack_advice)$dept <- atributos$DEPT
krack_advice
## IGRAPH DN-- 21 190 --
## + attr: name (v/c), age (v/n), tenure (v/n), level (v/n), dept
## | (v/n)
## + edges (vertex names):
## [1] X1->X2 X1->X4 X1->X8 X1->X16 X1->X18 X1->X21 X2->X6 X2->X7
## [9] X2->X21 X3->X1 X3->X2 X3->X4 X3->X6 X3->X7 X3->X8 X3->X9
## [17] X3->X10 X3->X11 X3->X12 X3->X14 X3->X17 X3->X18 X3->X20 X3->X21
## [25] X4->X1 X4->X2 X4->X6 X4->X8 X4->X10 X4->X11 X4->X12 X4->X16
## [33] X4->X17 X4->X18 X4->X20 X4->X21 X5->X1 X5->X2 X5->X6 X5->X7
## [41] X5->X8 X5->X10 X5->X11 X5->X13 X5->X14 X5->X16 X5->X17 X5->X18
## [49] X5->X19 X5->X20 X5->X21 X6->X21 X7->X2 X7->X6 X7->X11 X7->X12
## + ... omitted several edges
No post 01 já mostramos como podemos usar os atributos para plotar as redes. Recomendo que experimentemos várias combinações de acordo com o que pretendemos investigar.
Visualizações interativas com tkplot
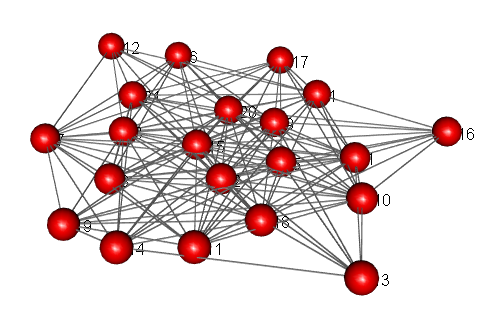
Podemos gerar visualizações no R com o comando tkplot:
tkplot(krack_advice, vertex.label.cex=.8, vertex.label.color="black", vertex.color="red", edge.arrow.size=0.2)
## [1] 1
Essa visualização nos permite editar de forma interativa alguns elementos do grafo (excetuando-se tamanho e cor dos vértices e laços que devem ser preestabelecidos). Se conseguirmos obter um resultado visual satisfatório e elegante, é bom que façamos a extração das coordenadas para exportamos para outros softwares, caso seja necessário. Para isso, usamos o comando tk_coords() com o primeiro parâmetro sendo o id do grafo gerado no output do comando anterior.
coordenadas <- tk_coords(1, norm=T)
A partir dessas coordenadas, podemos ainda gerar outro gráfico pelo método convencional mas com a localização dos vértices que gostamos.
plot(krack_advice, vertex.label.cex=.8, vertex.label.color="black", vertex.color="red", edge.arrow.size=0.2, layout=coordenadas, main="Krackhardt ADVICE")

Visualizações em 3D
O R gera algumas visualizações em 3D bastante elegantes. Para um grafo 3D, faça
rglplot(krack_advice, vertex.label.cex=.8, vertex.label.color="black", vertex.color="red", edge.arrow.size=0.2, layout=layout_with_kk)
Para salvar o grafo 3D em uma imagem, use
snapshot3d("rede3d.png", "png", top=F)
Aqui está um exemplo
Outra opção interessante é trabalhar com o pacote networkD3. Para isso, faça
library(networkD3)
krack3d <- igraph_to_networkD3(krack_advice)
simpleNetwork(krack3d$links, Source="source", Target="target", fontSize = 10)
Para um exemplo de gráfico em 3D com o pacote networkD3 com a rede de advogados de Lazega, acesse aqui. Aproveito para agradecer à querida Bruna Wunderwald pela dica deste pacote. Valeu, Bruna!
Redes 2-mode
Para trabalhar com redes 2-mode no R é preciso saber que o pacote igraph “entende” uma rede de 2 modos quando ela tem um atributo chamado type com valores lógicos TRUE e FALSE. Para usarmos um exemplo, vamos importar a rede de membros da Academy of Management (AOM Membership). O procedimento no UCINET já está descrito acima. Para importar, faça
aom <- read.csv2("C:/Users/.../UCINET data/AOM division membership.txt", header = T, stringsAsFactors = F)
aom <- as.matrix(aom)
Para importar a matriz de afiliações, use
aom <- graph_from_incidence_matrix(aom)
is.bipartite(aom)
## [1] TRUE
summary(V(aom)$type)
## Mode FALSE TRUE NA's
## logical 3324 23 0
Quando temos uma edge list, formato muito comum de dados relacionais, o procedimento requer alguns detalhes a mais. Depois de abrir a edge list e transformá-la num objeto igraph, fazemos um mapeamento dos vértices que pertencem a cada modo para gerar o grafo 2-mode. Depois, salvamos o mapeamento para cada vértice em um objeto (que é uma lista) e atribuímo-lo a um novo atributo criado com o nome type. Para realizar esta tarefa, usarei uma rede de parlamentares em comissões coletada usando o pacote bRasilLegis. O código para baixar esses dados está neste outro post.
parlamentares <- read.csv2("C:/Users/.../orgaos.csv", stringsAsFactors = F)
parlamentares <- as.matrix(parlamentares)
parlamentares <- graph_from_edgelist(parlamentares, directed = F)
is.bipartite(parlamentares)
## [1] FALSE
type <- bipartite_mapping(parlamentares)
V(parlamentares)$type <- type[[2]]
is.bipartite(parlamentares)
## [1] TRUE
plot(parlamentares, vertex.label=NA, vertex.size=3, vertex.color=as.numeric(V(parlamentares)$type)+3, main="Rede de afiliações de \nParlamentares a comissões")

Sintam-se à vontade para comentar. Um abraço a todos.